- de Change Region
- Global Site
- Home
- Anwendungen
- Biowissenschaften
- Mikroskopie im Nanometer-Bereich
Mikroskopie im Nanometer-Bereich
Hunderte von Jahren war die Auflösung der Lichtmikroskopie grundsätzlich durch die Beugung begrenzt, wobei ~200 Nanometer (nm) als ungefähre Grenze in XY angesehen wurden. Dies mag ausreichen, um einzelne Zellen und viele subzelluläre Strukturen zu beobachten, aber es reicht nicht aus, um feine Details im Suborganellenmaßstab und einzelne Biomoleküle aufzulösen, die meistens Dimensionen im Bereich unter 200 Nanometer haben.
Die jüngste Geschichte war Zeuge der Erfindung mehrerer optischer Bildgebungsverfahren mit „Super-Auflösung“, die verschiedene Strategien zur Umgehung bzw. Durchbrechung der durch Beugung auferlegten Grenze ausnutzten. Ihre Bedeutung wurde mit dem Nobelpreis für Chemie im Jahr 2014 gewürdigt. Die Super-Resolution-Mikroskopie hat bereits zu mehreren Entdeckungen beigetragen, wie beispielsweise der periodischen axonalen Gitterstrukturen (axonal periodic scaffold).
Produkte für die Mikroskopie im Nanometer-Bereich
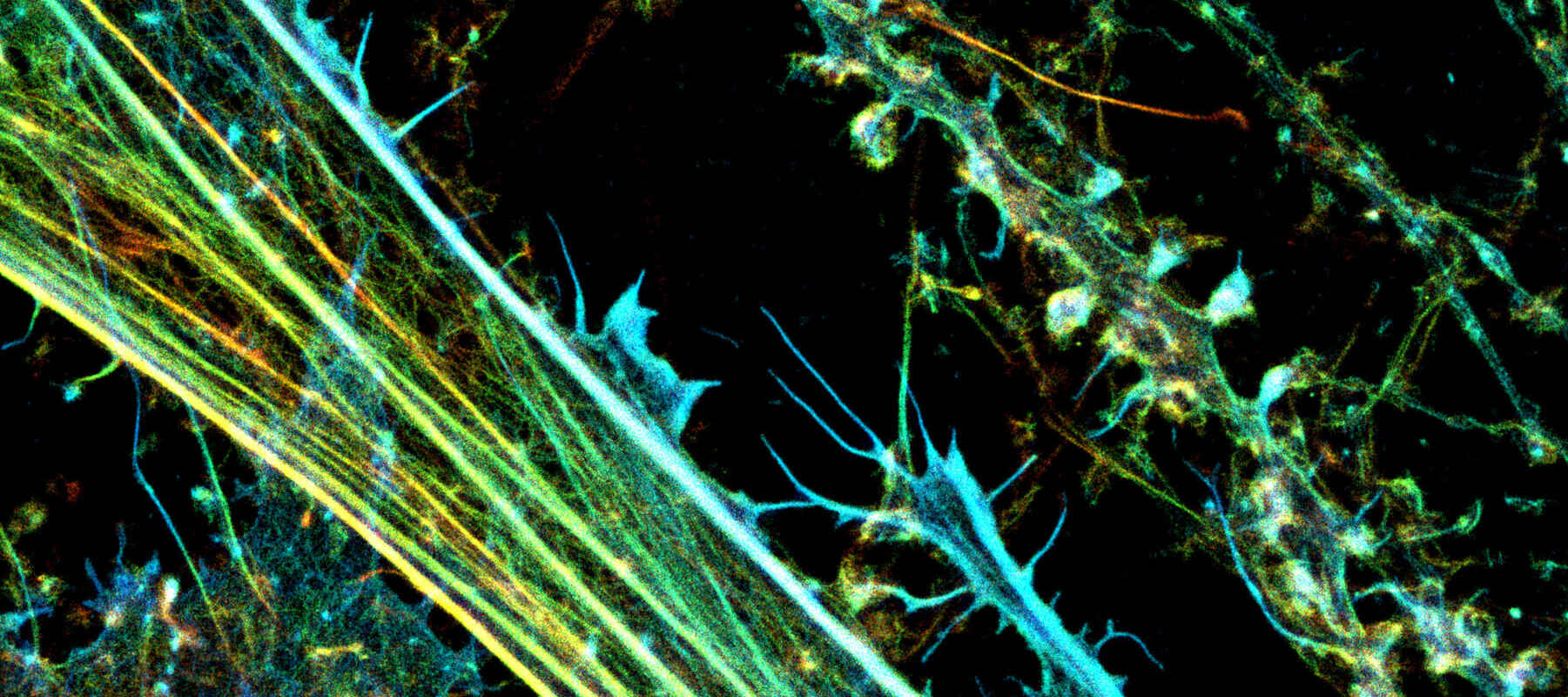
3D-STORM-Bild. Kultivierte Neurone mit fluoreszierend markiertem Aktin (Farbcodierte Z-Ebenen), aufgenommen mit einem Nikon N-STORM-System von Prof. Christophe Leterrier (CR1 CNRS).
Das Nikon N-STORM Super-Resolution Mikroskop-System bietet die höchste Auflösung der von Nikon angebotenen Systeme. Sie ist in XYZ etwa eine Größenordnung höher als die bei normaler Weitfeld-Epifluoreszenz. Das N-STORM ist auf dem inversen Mikroskop Ti2-E mit dem Hardware-basierten Fokus-Stabilitäts-System Perfect Focus System 4 (PFS4) konfiguriert. Damit ist auch ein extrem feines Durchfokussieren im Z-Stapel-STORM-Aufnahmemodus möglich, wodurch die außerordentlich hohe Stabilität beim Imaging kleinster Volumina mit einer Dicke von bis zu ~5 µm gewährleistet ist. Mit dem N-STORM-Illuminator ist auch konventionelle TIRF-Mikroskopie möglich..
Das N-SIM S Super-Resolution-Mikroskop-System verwendet für die Beleuchtung mit Gittermuster einen räumlichen Lichtmodulator. Dieser bewerkstelligt die notwendigen Richtungs- und Orientierungswechsel der Gitterbeleuchtung und damit der Interferenzmuster in extrem schneller Abfolge. Dadurch erhält die Aufnahmegeschwindigkeit einen enormen Schub, sowohl für die 9 Bilder im 2D- und TIRF- als auch für die 15 Bilder im 3D-SIM-Modus. Im 9-Bild-SIM-Modus kommt man auf die beachtlichen Raten von bis zu 15 Bildern pro Sekunde (FPS) – das ist kompatibel mit vielen Live-Cell-Imaging-Anwendungen. TIRF-SIM bietet zusätzlich die Erhöhung der Auflösung in XY, zusammen mit dem eigentlichen „optical sectioning“-Effekt der TIRF-Technik. Das N-SIM E Super-Resolution-Mikroskop- System ist ein kostengünstigeres SIM-System für 3D-SIM-Imaging und bietet die gleiche Auflösungsverbesserung wie das N-SIM S.
Das Yokogawa CSU-W1 SoRa Super-Resolution-System ist ein konfokales Spinning-Disk-Mikroskop, dessen Emissions-Disk mit Mikrolinsen die Superauflösung durch optisches Pixel-Reassignment realisiert. Das Spinning-Disk-Design des CSU-W1 SoRa macht die Bildaufnahme mit Super-Resolution vergleichsweise schnell.
●: enthalten, ⚬: option
| N-STORM Super-Resolution-Mikroskopsystem |
N-SIM S Super-Resolution-Mikroskopsystem |
N-SIM E Super-Resolution-Mikroskopsystem |
Yokogawa CSU-W1 SoRa Spinning Disk Einheit für Super-Resolution-Imaging |
|
|---|---|---|---|---|
| Optische Auflösungsgrenzen | ~20 nm (XY)* ~50 nm (Z)* |
~115 nm (XY)* ~86 nm (XY; TIRF-SIM-Modus)* ~269 nm (Z; 3D-SIM-Modus)* |
~115 nm (XY)* ~269 nm (Z; 3D-SIM-Modus)* |
~120 nm (XY)* |
| Grenzbereich für das Imaging in tieferen Schichten | ~ 5 μm | ~ 10 – 20 μm | ~ 10 – 20 μm | ~ 50 – 100 μm |
| Bildaufnahmeraten | ~0.1 FPS (bis 500 Hz Aufnahme von Einzelbildern) |
~15 FPS (nur 2D-SIM und TIRF-SIM) |
~1 FPS (alle Modi) |
~30+ FPS (nur limitiert durch das Signal-Rausch-Verhältnis, die Rotationsgeschwindigkeit der Spinning-Disk und die Ausleserate der verwendeten Kamera) |
| Spektralkanäle | 3 | 6 | 3 | 6 |
| Maximales Gesichtsfeld (Imaging area) | 80 x 80 μm (mit 100X Objektiv) |
66 x 66 μm (mit 100X Objektiv) |
66 x 66 μm (mit 100X Objektiv) |
61 x 57 μm (unter Verwendung eines 100-fachen Objektivs mit 2,8-facher Nachvergrößerung) |
| Kompatible Mikroskopstative | N-STORM | N-SIM S | N-SIM E | CSU-W1 SoRa |
| ECLIPSE Ti2-E Invertiertes Forschungsmikroskop | yes | yes | yes | yes |
| ECLIPSE Ti2-A Invertiertes Forschungsmikroskop | no | no | no | yes |
| ECLIPSE Ti2-U Invertiertes Forschungsmikroskop | no | no | no | yes |
*Diese Werte dienen als ungefähre Anhaltspunkte. Die Auflösungsleistung hängt von den jeweils individuellen Bedingungen ab. Für Dimensionen, bei denen keine Verbesserung erwartet wird, werden keine Auflösungsschätzungen angegeben.
Zugehörige Literatur
Diskussion: Mikroskopie im Nanometer-Bereich
So finden Sie die Super-Resolution-Methode, die für Sie richtig ist
Quantitative Zeitraffer-Analyse einzelner Spines mit Z-Stapel-Projektionen mit 3D-SIM,
Belichtungszeit 100 ms, Zeitintervalle 120 s
Zeitraffer-Faktor 11 Mal
Anregungswellenlänge 488 nm
Probe: GFP exprimierendes Hippocampus-Neuron (Maus), dendritische Spines
Movie mit freundlicher Genehmigung von: Drs. Yutaro Kashiwagi und Shigeo Okabe, Department of Cellular Neurobiology, Graduate School of Medicine and Faculty of Medicine, The University of Tokyo.
Die Super-Resolution-Mikroskopie bietet eine Auflösungsverbesserung über die Beugungsgrenze hinaus, aber jede Technik hat auch ihre jeweiligen Einschränkungen und Kompromisse, die gut verstanden werden sollten, um festzustellen, welche Technik für Ihre Forschungsaufgaben am besten geeignet ist.
Zuallererst sind die Bildaufnahmeraten bei Super-Resolution im Allgemeinen langsamer als bei herkömmlichen Techniken. Das Imaging mit N-STORM ist für die Anwendung in lebenden Zellen aufgrund der vergleichsweise langsameren Aufnahmerate (in der Regel mehr als eine Sekunde pro Bild) schwierig. STORM und andere SMLM-Techniken bieten die größte Auflösungsverbesserung, sind jedoch mit einer begrenzten Anzahl von Fluorophoren kompatibel, von denen viele spezielle Puffersysteme erfordern, die oft nicht mit lebenden Zellen kompatibel sind.
Das N-SIM S und das Yokogawa CSU-W1-SoRa eignen sich besser für superauflösendes Imaging lebender Zellen. Beide Systeme unterstützen schnellere Bildgebungsraten (15 FPS mit dem N-SIM S, noch höher mit dem CSU-W1-SoRa) und können mit herkömmlichen Fluorophoren sowie ohne spezifische Puffersysteme verwendet werden.
Hochleistungsobjektive für Super-Resolution-Mikroskopie
Nikon-Objektiv mit automatisiertem Korrekturring, montiert an einem inversen Mikroskop Ti2-E.
Einer der kritischsten Faktoren im Hinblick auf die Maximierung der optischen Auflösung ist die Wahl des Objektivs. Dies gilt gleichermaßen für die Super-Resolution wie für alle anderen Mikroskopier-Techniken.
Die numerische Apertur (NA) des Objektivs sollte möglichst hoch sein, wobei auch immer ein geeignetes Immersionsmedium verwendet werden sollte. Die Nikon Objektiv-Reihe Super-Resolution (SR) umfasst das CFI SR HP Apochromat TIRF 100XC Oil (Ölimmersion, NA = 1,49), das CFI SR HP Plan Apochromat Lambda S 100XC Sil (Silikonimmersion, NA = 1,35) und das CFI SR Plan Apochromat IR 60XC WI (Wasserimmersion, NA = 1,27). Diese Objektive bieten einige der höchsten numerischen Aperturen (NA) in ihren jeweiligen Klassen.
Ausgewählte Objektive der Super-Resolution-Serie sind mit einem motorisierten, automatischen Korrekturring erhältlich, der - ohne manuelles Eingreifen - eine Feinabstimmung der sphärischen Aberrationskorrektur ermöglicht und so die höchstmögliche Auflösung und 3D-Imaging-Leistung gewährleistet.
Verbesserte Auflösung – Überwindung der Grenzen traditioneller Technologien
Konfokale Bilder (Point-Scanner). Cilien von Cochlea-Zellen-. XY- und XZ-Ansichten mit (rechts) und ohne (links) Anwendung der Einstellungen für eine erhöhte Auflösung.
Manchmal liegt die zur Beantwortung Ihrer Frage erforderliche Auflösung nicht zu weit weg von der konventioneller Techniken und erfordert daher möglicherweise auch nicht den einen oder anderen Kompromiss, den Super-Resolution-Techniken manchmal abverlangen. In einem solchen Fall empfehlen wir Ihnen, das Potenzial dessen zu berücksichtigen, was wir als konfokale Mikroskopie mit „verbesserter Auflösung“ bezeichnen.
Was ist konfokale Mikroskopie mit verbesserter Auflösung? Der wichtigste Punkt, den es zu verstehen gilt, ist, dass das traditionelle konfokale Mikroskop bereits zu einem gewissen Grad eine Auflösungsverbesserung leistet, die über das hinausgeht, was mit einem normalen Weitfeldmikroskop möglich ist; lateral (XY) ist die theoretische Grenze bei ~140 nm (XY) gut beschrieben. Das Erreichen dieser Grenze erfordert jedoch die Verwendung einer unendlich kleinen Lochblende. Glücklicherweise kann in der Praxis eine Pinhole-Größe von ~0,5 Airy –Einheiten mit einer iterativen 3D-Dekonvolution nach der Aufnahme kombiniert werden, um sich dieser Grenze zu nähern, wobei eine Auflösung von ~160 nm (XY) einen realistischen Wert für eine Reihe von Bedingungen beim Imaging darstellt.
Die konfokalen Point-Scanning Mikroskope AX / AX R von Nikon ermöglichen ein Imaging mit verbesserter Auflösung. Dazu kommen die Vorteile, die Feineinstellungen für die meisten Fluorophore im sichtbaren Spektrum sehr genau abzustimmen und gleichzeitig bis zu vier Spektralkanäle aufzunehmen. Die Lochblende (pinhole) ist stufenlos verstellbar und hat eine sechseckige (anstatt quadratische) Form, wodurch eine präzisere konfokale Abbildung ermöglicht wird. Das konfokale Mikroskop AX R mit Resonanz-Scanner ermöglicht es, Bilder mit Videorate (30 FPS) und schneller aufzunehmen und gleichzeitig von der verbesserten Auflösung zu profitieren.
Glossar
- Bildaufnahmeraten
- Die Bildaufnahmeraten werden in Bildern pro Sekunde (FPS) angezeigt.
- Grenzbereich für das Imaging in tieferen Schichten
- Das ist die ungefähre Distanz entlang der Z-Achse, über die das jeweilige System mit dem Objektiv in tiefere Schichten der Probe „Hineinfokussieren“ kann, um noch optische Schnitt-Bilder mit angemessener Qualität und ausreichendem Signal-Rausch-Verhältnis zu liefern. Dieser Wert kann sehr variabel sein und hängt stark von den optischen Eigenschaften der Probe, der Art der Probenkammer sowie der (Fluoreszenz-) Färbung ab.
- Kompatible Mikroskopstative
- Dies bezieht sich auf die Nikon-Mikroskopstativmodelle, die mit jedem System kompatibel sind.
- Maximales Gesichtsfeld (Imaging area)
- Das größte Gesichtsfeld (im Präparat gemessen), das die gegebene Technik beim Vergrößerungsfaktor des angegebenen Objektivs ermöglicht.
- Optische Auflösungsgrenzen
- Die jeweiligen optischen Auflösungsgrenzen des gegebenen Systems. Der Wert für die laterale (XY) Auflösung unterscheidet sich typischerweise von dem für die axiale (Z) Auflösung. Wenn kein Z-Auflösungswert angegeben ist, gibt es in dieser Dimension keine Verbesserung.
- Spektralkanäle
- Dies bezieht sich auf die Anzahl möglicher spektraler (Farb-) Kanäle, die unterstützt werden (unter der Annahme einer Laserlinie für jeden Fluoreszenzfarbstoff).
- Home
- Anwendungen
- Biowissenschaften
- Mikroskopie im Nanometer-Bereich