- de Change Region
- Global Site
- Home
- Anwendungen
- Biowissenschaften
- Live-Cell-Imaging
Live-Cell-Imaging
Für die moderne zellbiologische Forschung ist die Bedeutung der Lebendzellmikroskopie – insbesondere in Verbindung mit der Fluoreszenz-Mikroskopie – nicht hoch genug einzuschätzen. Mit Live Cell Imaging und Fluoreszenz-Methoden lassen sich Dynamik und Wechselwirkungen spezifischer Biomoleküle über längere Zeiträume visualisieren, aufzeichnen und analysieren. In den letzten Jahrzehnten haben zahlreiche technologische Innovationen die Einsatzmöglichkeiten des Live Cell Imaging stark erweitert. Dazu beigetragen hat die Entwicklung von fluoreszierenden Proteinen als genetisch exprimierbare und spezifische Marker. Die Mikroskopie lebender Zellen wird typischerweise auf einem inversen Mikroskop durchgeführt, auf dem die mit Medium gefüllten Kultur-Gefäße und -Kammern besser montiert werden können.
Produkte für Live Cell Imaging
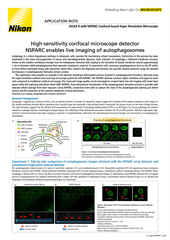
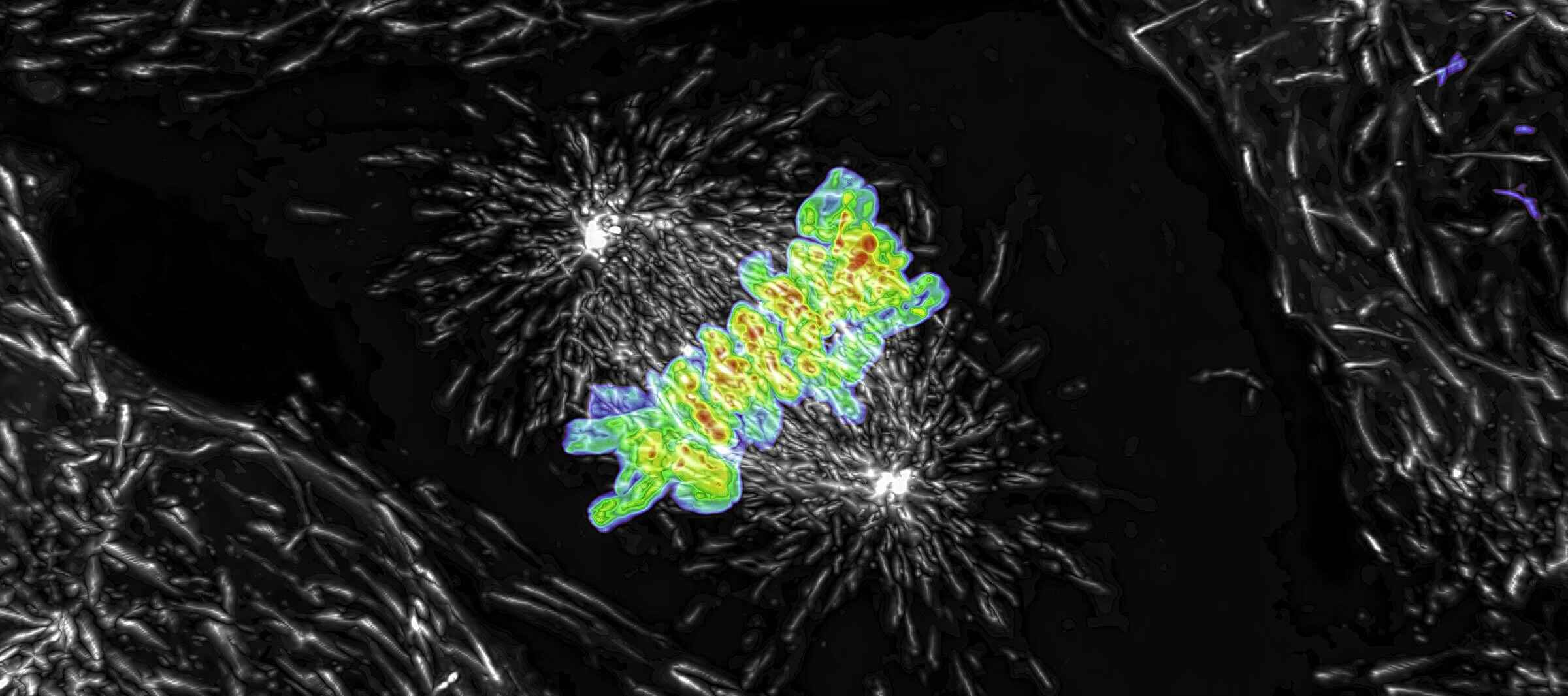
Projektion eines Z-Bildstapels (Maximum Intensität), aufgenommen von einer lebenden mitotischen Zelle mit dem AX R-Resonanz-Scanner mit 15 FPS und einer Pixelauflösung von 2048 x 1024. Objektiv CFI Plan Apo Lambda 60X Öl NA 1.4.
Das motorisierte inverse Mikroskop ECLIPSE Ti2-E ist das universellste Mikroskopstativ für das Live Cell Imaging – es kann mit allen hier beschriebenen konfokalen und Super-Resolution Techniken optional kombiniert werden. Das Perfect Focus System 4 (PFS4) stellt die weltweit führende hardwarebasierte Technologie für zuverlässiges Halten des Fokus zur Verfügung, die Langzeit-Live Cell Imaging ohne Fokusdrift ermöglicht und sowohl mit Glas- als auch mit Kunststoffkulturgefäßen kompatibel ist. Ein automatisierter Spender für das Wasser für Wasserimmersionsobjektive ist ebenfalls erhältlich.
Das modulare Beleuchtungssystem Ti2-LAPP ermöglicht die Kombination von bis zu fünf verschiedenen Beleuchtungsmodulen in einem einzigen Mikroskop, wobei Optionen für TIRF, Weitfeld-Epifluoreszenz und Photostimulation bestehen, einschließlich Baugruppen für FRAP, sowie Illuminatoren mit digitalen Mikrospiegel-Arrays (DMDs). Es ist möglich, mehrere TIRF-Beleuchtungsmodule in ein einziges Stativ zu integrieren, wodurch simultanes Mehrkanal-Imaging mit jeweils optimalem TIRF-Winkel für jede Laserlinie ermöglicht wird.
Die konfokalen Mikroskope AX / AX R sind die Point-Scanner-Systeme von Nikon – wobei das AX R über einen schnellen Resonanz-Scanner verfügt, der konfokale Mikroskopie mit Videorate von 30 Bildern pro Sekunde (FPS) bei 512 x 512 Pixel unterstützt. Das Softwaremodul Denoise.ai entfernt automatisch das sog. Schrotrauschen – die Hauptursache für Rauschen in konfokalen Bildern mit Resonanz-Scanning.
Die konfokalen Spinning-Disk-Systeme der CSU-Serie von Yokogawa sind beliebte Lösungen für das Imaging lebender Zellen. Das CSU-W1 eignet sich besser für das Imaging dickerer Präparate, wie z. B. Organoide. Der größere Abstand zwischen den Lochblenden, der das Crosstalking zwischen benachbarten Lochblenden verringert ermöglicht es, aus tieferen Schichten in streuenden Proben gute konfokale Bilder zu erzeugen.
Die Super-Resolution-Mikroskopie wird für Live-Cell-Imaging-Anwendungen immer mehr verfeinert und praktikabler. Die Emissionsscheibe des konfokalen Yokogawa CSU-W1 SoRa Spinning-Disk-Systems besitzt Mikrolinsen, um Super-Auflösung durch optisches Pixel-Reassignment zu realisieren.
●: enthalten, ⚬: option
| ECLIPSE Ti2-E Inverses Mikroskop (Weitfeld-Mikroskopie)* |
AX R Konfokales Mikroskop mit Resonanz-Scanner |
CSU-X1 Konfokales Mikroskop mit Spinning-Disk |
CSU-W1 Konfokales Mikroskop mit Spinning-Disk |
Ti2-LAPP E-TIRF Illuminator |
N-SIM S Super-Resolution-Mikroskop |
CSU-W1 SoRa Konfokales und Super-Resolution-Mikroskop mit Spinning-Disk |
|
|---|---|---|---|---|---|---|---|
| Grenzbereich für das Imaging in tieferen Schichten | ~ 5 μm ~ 15 – 25 μm (mit Dekonvolution) |
~ 100 – 500 μm | ~ 50 μm | ~ 50 – 100 μm | ~ 100 – 300 nm | ~ 10 – 20 μm | ~ 50 – 100 μm |
| Unterstützt Imaging mit Video-Rate | Ja |
Ja (30 FPS bei 512 x 512 Pixel Scan) |
Ja |
Ja (limitiert durch das Kamerasystem und die Rotationsgeschwindigkeit der Spinning Disk) |
Ja (limitiert durch das Kamerasystem) |
Nein (15 FPS nur mit 2D-SIM und TIRF-SIM) |
Ja (limitiert durch das Kamerasystem und die Rotationsgeschwindigkeit der Spinning Disk) |
| Sehfeld | 25 mm Diagonale (rund) | 25 mm Diagonale (quadratisch) | 10 x 7 mm (rechteckig) | 17 x 16 mm (rechteckig) | ~ 10 mm Diagonale (rund) | ~ 9.3 mm Diagonale (rund) | 17 x 16 mm (rechteckig) |
| Unterstützte Mikroskopiertechniken | ECLIPSE Ti2-E | AX R | CSU-X1 | CSU-W1 | Ti2-LAPP E-TIRF | N-SIM S | CSU-W1 SoRa |
| Hellfeld | yes | no | no | no | no | no | no |
| Konfokal – Punkt-Scanner | no | yes | no | no | no | no | no |
| Konfokal – Spinning Disk | no | no | yes | yes | no | no | yes |
| Dunkelfeld | yes | no | no | no | no | no | no |
| Differentieller Interferenzkontrast (DIC) | yes | no | no | no | no | no | no |
| Nikon Advanced Modulation Contrast (NAMC) | yes | no | no | no | no | no | no |
| Phasenkontrast | yes | no | no | no | no | no | no |
| Super-Resolution - Strukturierte Beleuchtung | no | no | no | no | no | 2D-SIM, TIRF-SIM, 3D-SIM | |
| Super-Resolution - Optisches Pixel-Reassignment | no | no | no | no | no | no | yes |
| Total Internal Reflection Fluoreszenz (TIRF) | no | no | no | no | yes | no | no |
| Volumenkontrast | yes | no | no | no | no | no | no |
| Weitfeld-Fluoreszenz | yes | no | no | no | no | no | no |
| Kompatible Mikroskopstative | ECLIPSE Ti2-E | AX R | CSU-X1 | CSU-W1 | Ti2-LAPP E-TIRF | N-SIM S | CSU-W1 SoRa |
| ECLIPSE Ti2-E invers | no | yes | yes | yes | yes | yes | yes |
| ECLIPSE Ti2-A invers | no | no | yes | yes | no | no | yes |
| ECLIPSE Ti2-U invers | no | no | yes | yes | no | no | yes |
| ECLIPSE Ni-E aufrecht | no | yes | yes | yes | no | no | no |
| ECLIPSE Ni-U aufrecht | no | no | yes | yes | no | no | no |
| ECLIPSE FN1 aufrecht | no | yes | yes | yes | no | no | no |
*Diese Spalte bezieht sich ausschließlich auf die Verwendung des ECLIPSE Ti2-E für Weitfeld-Mikroskopie. Das Ti2-E ist das Mikroskopstativ, welches für die Verwendung zusammen mit den anderen Systemen in der Tabelle empfohlenen wird.
Zugehörige Literatur
Diskussion: Live Cell Imaging
„The Art and Science“ des Live Cell Imaging
Die Nikon Objektiv-Reihe mit Silikon- Immersion. Jedes dieser Objektive bietet eine hohe NA, einen großen Arbeitsabstand und einen Korrekturring mit temperaturabhängigen Einstellmöglichkeiten.
Die Lebendzellmikroskopie verlangt von der Mikroskopikerin und dem Mikroskopiker immer wieder, alle technischen Parameter für die Mikroskopie mit viel Gespür so sorgfältig auszubalancieren, dass zielgerichtet die wesentlichen Daten in der Art aufgezeichnet werden können, dass Störungen der lebenden Zellen so minimal wie möglich gehalten werden. Während es zum Beispiel sehr verlockend sein kann, mehr Beleuchtungsintensität, längere Kamerabelichtungen usw. zu verwenden, um Bilder mit einem besserem Signal-Rausch-Verhältnis (SNR) zu erhalten, ist es auf der anderen Seite wichtig zu verstehen, dass Phototoxizität die physiologische Integrität und die fortgesetzte Lebensfähigkeit der lebenden Proben mehr oder weniger stark beeinträchtigen kann. Darüber hinaus birgt das Photobleaching einen technischen Grenzwert dafür, wie viele Photonen pro Zeiteinheit im Verlauf des Intravital-Experiments aus jedem Fluorophor „verbraucht“ werden können.
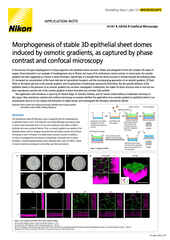
Ein weiterer ganz wichtiger Faktor ist die Wahl des Objektivs. Öl-Objektive mit hoher numerischer Apertur (NA) können sich gut für die Mikroskopie von Strukturen flacher Zellen in unmittelbarer Nähe zum Deckglas eignen, sie bringen jedoch sphärische Aberration mit sich, je tiefer man in die Probe hinein fokussiert. Dies ist zurückzuführen auf den relativ niedrigeren Brechungsindex (RI) der Zellen und des Kulturmediums im Vergleich zum RI von Glas und Öl. Für 3D-Imaging-Anwendungen und die Mikroskopie in tieferen Zell- und Gewebeschichten werden Objektive empfohlen, die mit Medien immergiert werden, die dem RI von Zellen und Medium besser entsprechen, wie z. B. Wasser oder Silikon.
Die Objektive CFI Apochromat Lambda S umfassen eine Reihe von Wasserimmersionsobjektiven, darunter das CFI Plan Apochromat IR 60XC WI, das mit NA = 1,27 die höchste NA für ein Wasserimmersionsobjektiv hat, die uns bekannt ist. Nikon hat vor kurzem seine Objektiv-Reihe mit Silikon-Immersion vorgestellt. Silikon hat einen Brechungsindex von etwa 1,4, der dem von lebenden Zellen und dem Medium besser entspricht, und dazu höhere NAs ermöglicht als für Wasserimmersionen. Diese Objektive eignen sich für die Mikroskopie von 3D-Kulturen wie beispielsweise Organoiden.
Das Minimieren von Phototoxizität und Photobleaching in lebenden Zellen
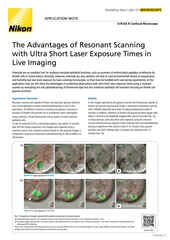
Das Triggern über die Hardware verkürzt erheblich die Gesamtzeit für die Imaging-Abfolge des jeweiligen Experiments. In den meisten dieser Beispiele war die Belichtungszeit der Kamera länger als die Auslesezeit der Kamera.
a Die Daten dieser letzten Zeile zeigen einen Vergleich, bei dem Belichtungs- und Auslesezeit der Kamera übereinstimmten und die Kamera im überlappten Modus (100 % Aussteuergrad (duty cycle)) betrieben werden konnte.
Die relativ intensive Beleuchtung, die in der Fluoreszenzmikroskopie eingesetzt wird, ist von Natur aus ungesund für lebende Zellen, insbesondere bei den energiereicheren Wellenlängen im Blaubereich. Aus diesem Grund wird oft die Verwendung von Rot- bis Nahinfrarot-Fluorophoren empfohlen, obwohl andererseits die Verwendung sehr populärer grün-fluoreszierender Fluorophore wie beispielsweise EGFP nicht gerade selten und insbesondere bei vielen Multicolor-Anwendungen gut funktioniert.
Zum Ansteuern von motorisierten Systemkomponenten verwenden die meisten Mikroskope das Triggern über die Software. Dies kann jedoch aufgrund der relativ ungenauen Computer-Hardwareuhr und der erforderlichen internen Zustands- Feedbacks und -Rückkopplungen das Gesamtsystem in unerwünschter Weise verlangsamen. Umgekehrt kann ein Triggern über die Hardware die hochgenaue Pixeluhr der Kamera nutzen, um motorisierte Komponenten koordiniert zu steuern, wobei die Notwendigkeit interner Zustand-Feedbacks umgangen wird. Dadurch wird die Systemgeschwindigkeit maximiert und folglich die Photonendosis auf die Proben minimiert. Zu triggernde Komponenten können Z-Piezotische, Laser-/LED-Illuminatoren und sogar kundenspezifische Geräte und Komponenten mit einer E/A-Kommunikationsfähigkeit sein.
Diese Art des Triggerns ermöglicht auch komplexere Regelkreise bzw. ein Definieren von Mustern für die Steuerung der Beleuchtung. Beispielsweise ist es über die Nikon NIS-Elements-Software möglich, die Probe nur dann der Beleuchtung auszusetzen, wenn alle Pixel einer sCMOS-Kamera belichtet werden, d.h. ein Fluoreszenzsignal erhalten (dabei nutzt man den Rolling-Shutter-Effekt der Kamera). Dadurch werden die Photonendosis und das Photobleaching im Vergleich zu einer Dauerbeleuchtung bei permanent laufender Bildaufnahme reduziert. Mit verschiedenen LED- und Laserbeleuchtungen ist es auch möglich, während der Belichtung eine Puls-Beleuchtung im Mikrosekundenbereich zu erzeugen. Durch eine Puls-Beleuchtung mit dieser Taktrate wird Fluorophoren, die in einen Triplett-Zustand angeregt wurden, zusätzliche Zeit gegeben, in den Grundzustand zu verfallen. Dies hilft, Hauptursachen für das Photobleaching zu vermeiden.
Die Verwendung von Deep Learning-basierter künstlicher Intelligenz zur Verbesserung der Lebendzell-Mikroskopie
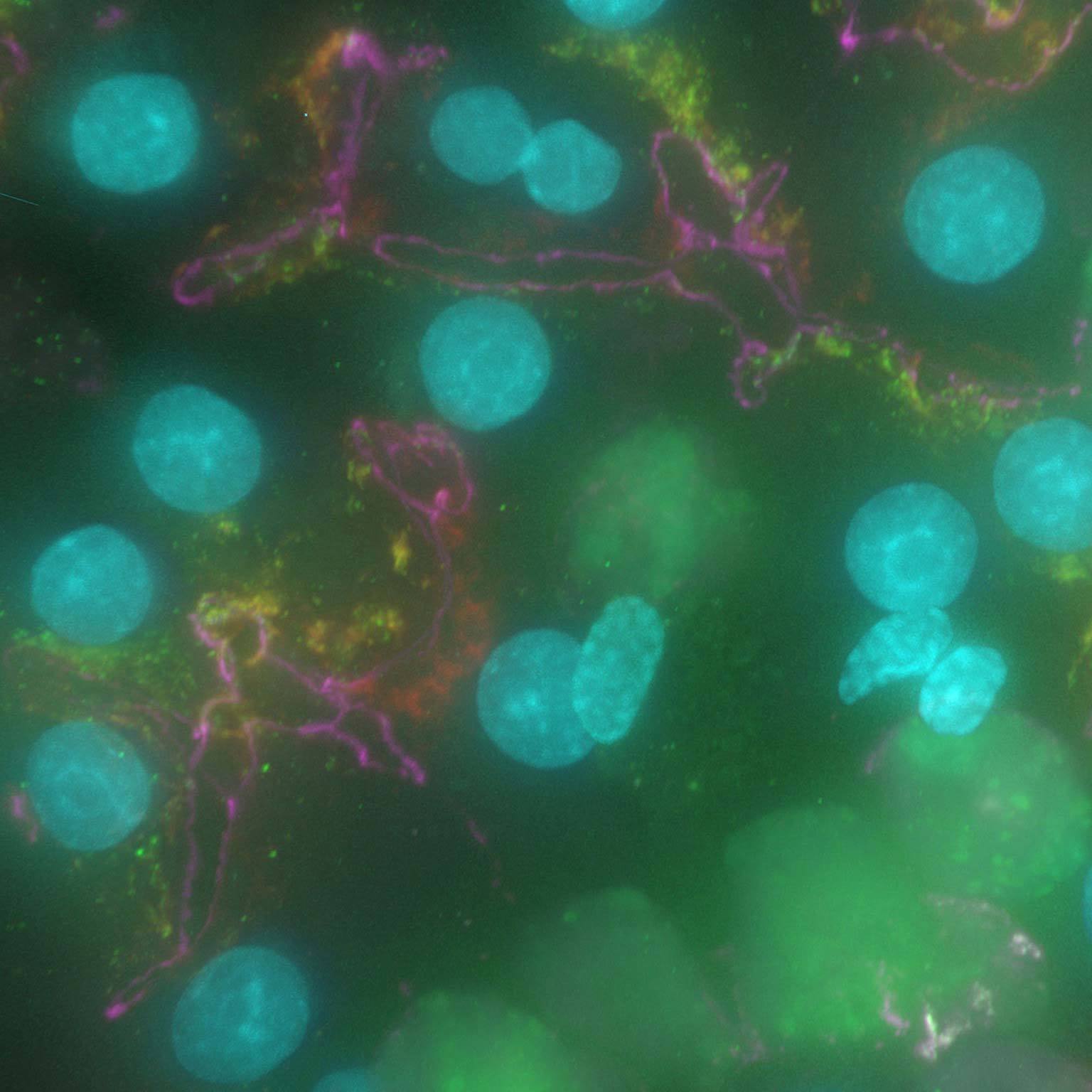
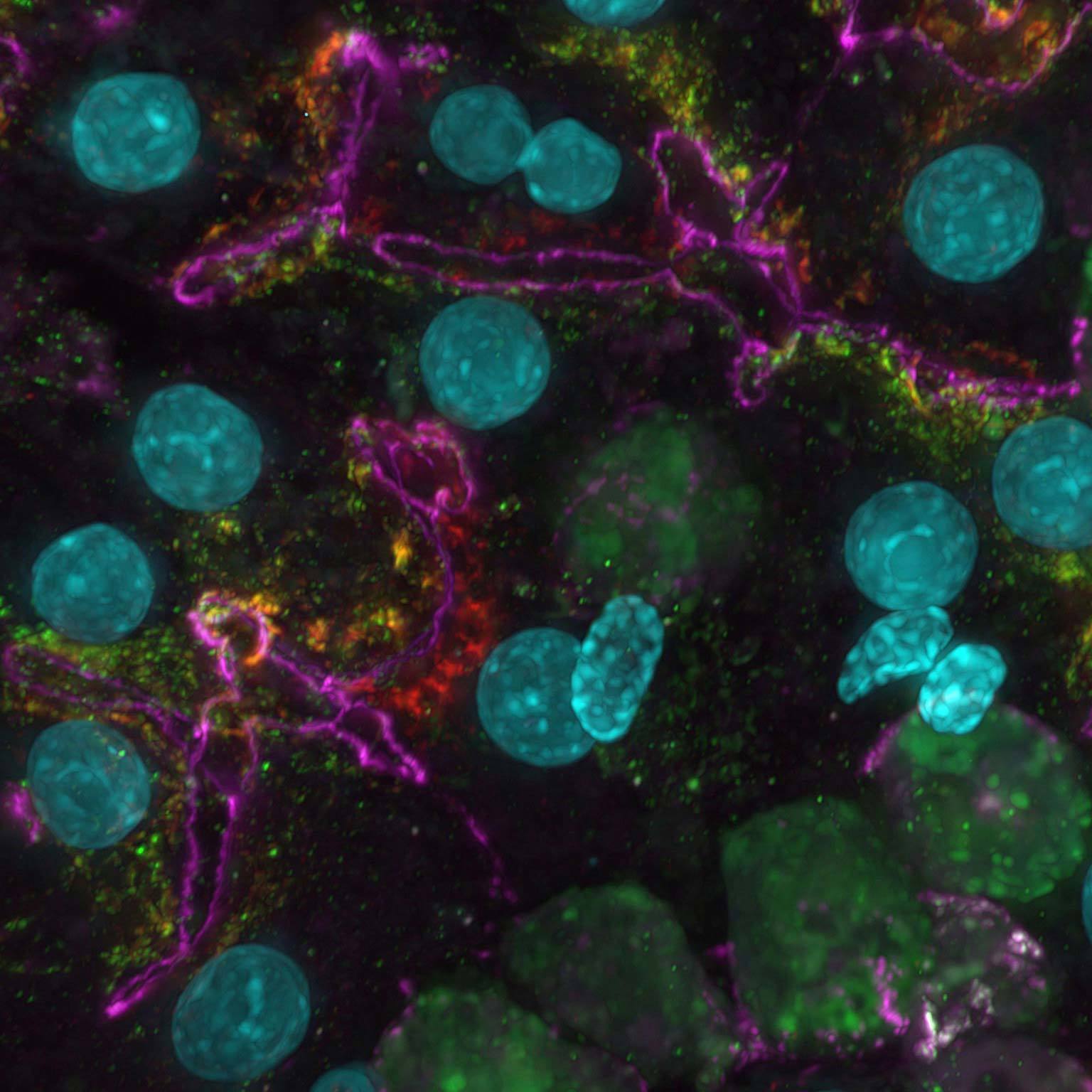
Bilder mit Weitfeld-Mikroskopie können durch Streulicht und unscharfe Bildsignale von ober- und unterhalb der Fokusebene "kontaminiert" sein. Mit AI-basierten Tools können aus den Rohbildern durch Herausrechnen der Rausch- und Überstrahlungsanteile Bilder mit sehr hohem Kontrast erzeugt werden.
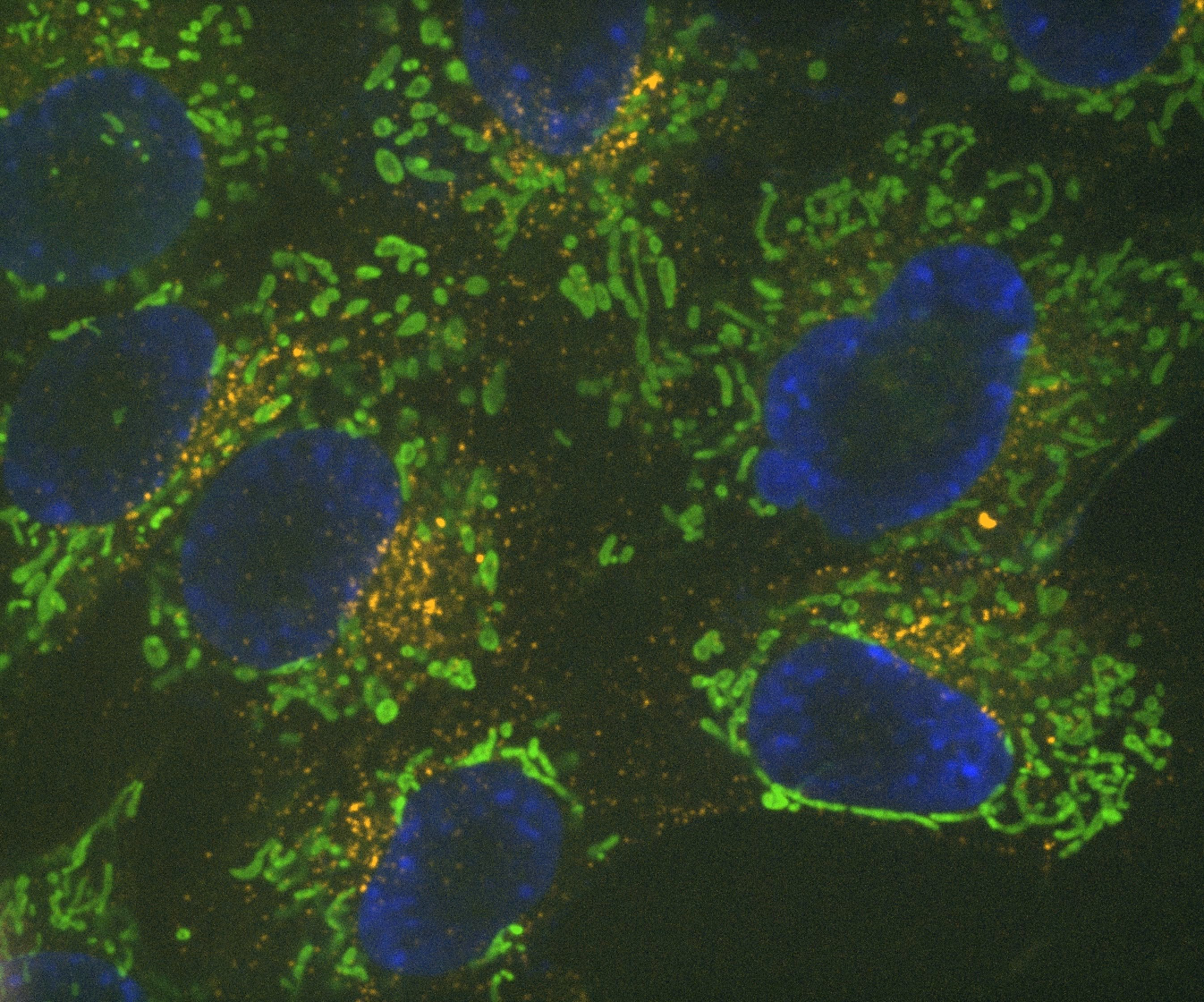
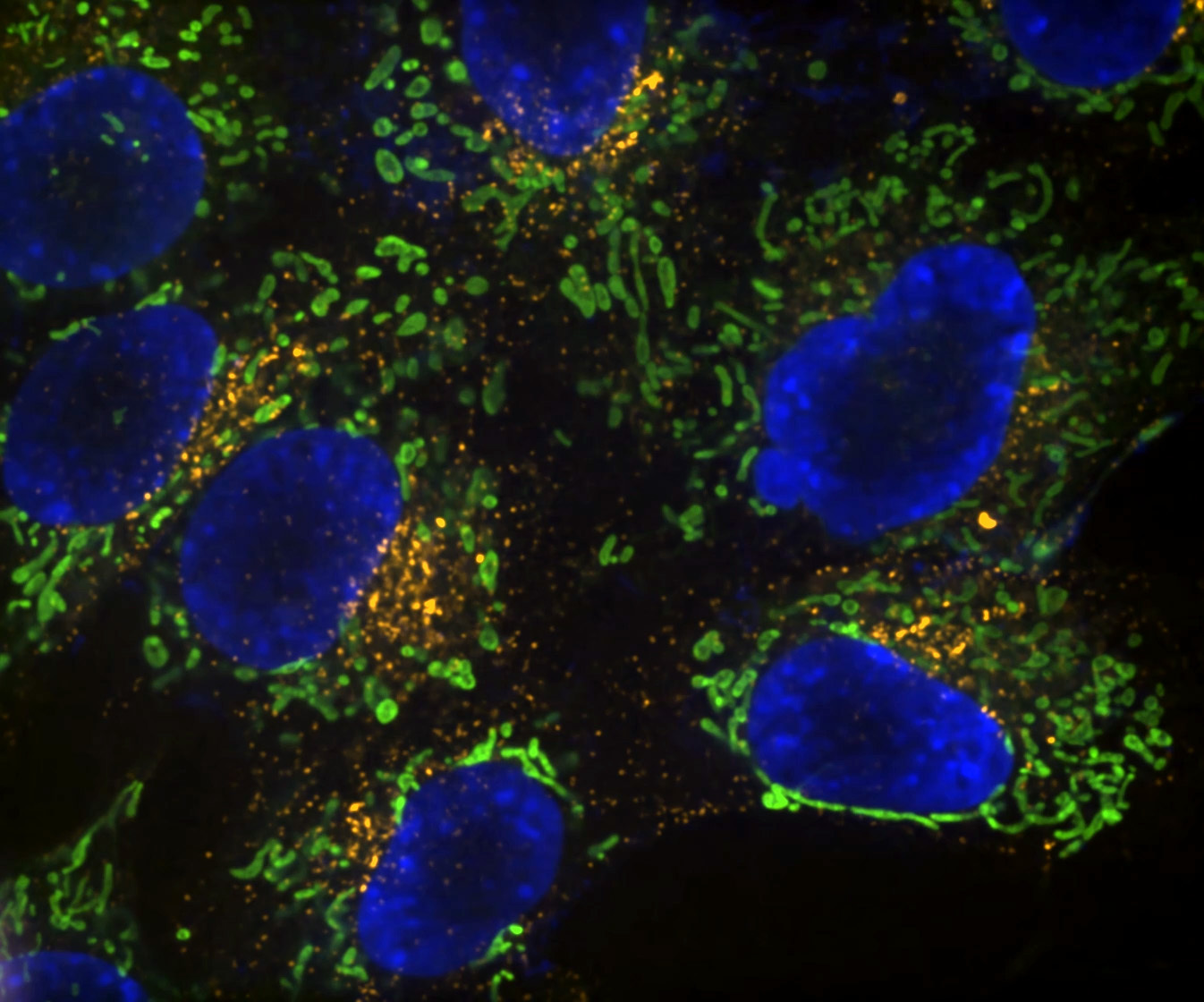
Denoise.ai erkennt und entfernt nur den Beitrag des Schrotrauschens in den Bildern, während die Signale der Strukturen und ihre relativen Intensitätswerte unberührt bleiben.
Das Aufkommen von Methoden der künstlichen Intelligenz (KI) für die Mikroskopie hat neue Möglichkeiten für das Live Cell Imaging eröffnet, die es auch schaffen, die Photonendosis weiter zu reduzieren. Deep Learning basierte Methoden zeigen ein hohes Potenzial für Assays, die auf Bildern von biologischen Proben basieren. Nikon hat sich der Entwicklung stabiler Softwarelösungen auf Basis von Deep Learning für verschiedene Anwendungen verschrieben, und stellt sie als NIS.ai-Module für seine NIS-Elements-Software zur Verfügung.
Clarify.ai entfernt aus normalen Weitfeld-Fluoreszenzbildern automatisch unscharfe Signale von außerhalb der Fokusebene. Dieses Softwaremodul ist von Nikon vortrainiert und erfordert seitens der Nutzer keine manuellen Anpassungen auf dem Weg zum Bildergebnis, dessen Beeinflussung durch User-Subjektivität („personal taste“) somit ausgeschlossen wird. Clarify.ai ermöglicht die unmittelbare Erstellung klarer, optischer Schnittbilder jeweils von der mit Weitfeldmikroskopie eingestellten Fokusebene, sodass die maximale Aufnahmegeschwindigkeit des Mikroskop-Systems ausgenutzt werden kann. Es ist nicht erforderlich - wie bei der iterativen 3D-Dekonvolution von Weitfelddaten –, Stapel von Bildern aus mehreren Ebenen zu generieren.
Denoise.ai ist ein weiteres vortrainiertes Modul und wird verwendet, um Poisson-Rauschen (Schrotrauschen) aus konfokalen Bildern in Echtzeit zu entfernen. Dies ermöglicht kürzere Belichtungszeiten und ist vorteilhaft bei Bildern, die mit dem Resonanz-Scanner des konfokalen Mikroskops AX R aufgenommen werden, wo das Schrotrauschen die Hauptursache für verrauschte Bilder ist.
Mit dem Nikon-Modul Convert.ai können Bildmerkmale vorhergesagt werden, die typischerweise mit Fluoreszenztechniken dargestellt werden. Die vorhergesagten Strukturen werden stattdessen aus entsprechenden Hellfeldbildern „herausgelesen“ (z. B. Vorhersage von DAPI-Kernfärbungsmustern aus DIC-Bilddaten). Dies erübrigt den Einsatz von Fluoreszenztechniken und vermeidet die vergleichsweise intensivere Beleuchtung. Hellfeld-Verfahren sind per se weniger phototoxisch.
Enhance.ai kann darauf trainiert werden, Strukturdetails, die in (Fluoreszenz-) Bildern mit niedrigem Signal-Rausch-Verhältnis (SNR) quasi „versteckt“ sind vorherzusagen und so darzustellen, als hätte ein exzellentes SNR bestanden. Auf diese Weise kann man Bilder mit schwachem SNR durch eine schonende Beleuchtungsintensität in Kauf nehmen, und den Schwerpunkt darauf legen, die lebenden Zellen bei reduzierter Photonendosis und möglichst wenig Photobleaching zu mikroskopieren.
Welche Mikroskopier-Technik ist die richtige für Ihre Forschung?
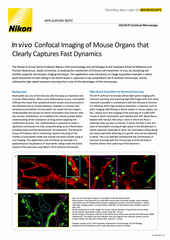
Weitfeld-Fluoreszenzbild eines Nierenglomerulus (Maus) in einem 16 mm dicken Gewebeschnitt vor (a) und nach (b) iterativer 3D-Dekonvolution mit der NIS-Elements-Software.
Für relativ flache, quasi zweidimensionale Proben, wie z. B. kultivierte adhärente Zellen, kann die Weitfeld-Fluoreszenzmikroskopie vollkommen ausreichend sein. Bei vielen Anwendungen und verschiedensten Probentypen kann die Weitfeld-Mikroskopie in Kombination mit Methoden der Dekonvolution oder der automatischen Entfernung von unscharfen Bildbestandteilen von außerhalb der Fokusebene z.B. mit dem Softwaremodul Clarify.ai ganz ausgezeichnete Ergebnisse bringen.
Konfokales Imaging wird notwendig, wenn Proben mit einer Dicke von mehr als ~20 μm mikroskopiert werden, es kann aber auch für die hochauflösende Darstellung von Proben mit einer Dicke von nur wenigen μm nützlich sind. Konfokale Spinning-Disk-Systeme sind vergleichsweise schnell und für Proben mit einer Dicke von bis zu ~50 μm geeignet (wobei das CSU-W1 aufgrund des größeren Pinhole-Abstands Bilder aus tieferen Schichten aufnehmen kann als andere Modelle). Beide Techniken sind für die räumliche Abbildung verschiedener Arten von 3D-Zellkultursystemen wie z.B. Organoiden und Sphäroiden anwendbar.
Konfokale Mikroskopie mit Point-Scannern kann aus tieferen Ebenen räumlicher Proben - bis hin zu mehreren hundert μm - optische Schnittbilder erzeugen. Der Resonanz-Scanner des konfokalen Systems AX R ist in der Lage, konfokale Bilder mit Videorate aufzunehmen und ist prädestiniert für die Kombination mit dem Softwaremodul Denoise.ai zur Echtzeit-Entfernung des Schrotrauschens. Im Gegensatz zu Spinning-Disk-Systemen verfügt das AX R über ein stufenlos einstellbares Pinhole zur Optimierung der Z-Ausdehnung der optischen Schnitte und der jeweiligen Auflösung eines jeden kompatiblen Objektivs.
Mit der Total Internal Reflection Fluorescence (TIRF)-Mikroskopie wird eine scharf begrenzte optische Beleuchtungsebene mit einer Dicke von wenigen hundert nm unmittelbar an der Grenzfläche zwischen Deckglas und Medium erzeugt, innerhalb der dort anhaftende Zellstrukturen exklusiv dargestellt werden können. Die TIRF-Beleuchtung sorgt für ein sehr gutes SNR und gestattet die Verwendung vergleichsweise geringer Laserleistung, was die Lebensfähigkeit von Zellen schützt und/oder die Bildaufnahmegeschwindigkeit maximieren lässt. Die Nikon Objektive-Reihe CFI Apochromat TIRF weist darüber hinaus eine sehr hohe NA von 1,49 auf.
Super-Resolution Mikroskopiertechniken werden eingesetzt, um Strukturdetails mit Dimensionen jenseits der Beugungsgrenze aufzulösen. Lebendzellmikroskopie mit Super-Resolution war im schon immer ziemlich herausfordernd aufgrund vieler Kompromisse, die von den verschiedenen Techniken verlangt wurden, um die Auflösung zu verbessern. Das Yokogawa CSU-W1 SoRa ist ein Super-Resolution-System, das vom Prinzip her ein konfokales Spinning-Disk-System ist und ein Imaging mit Geschwindigkeiten ermöglicht, die das CSU-W1 bereits schon schafft (vorausgesetzt, es ist ausreichendes Signal vorhanden).
Fluoreszenzmikroskopische Techniken besitzen ihre wesentliche Stärke durch ihre vielfältigen Möglichkeiten der spezifischen, molekularen Erkennung verschiedener Ziel-Moleküle und -Strukturen. Demgegenüber liefern Hellfeld-Techniken wie Phasenkontrast und differentieller Interferenzkontrast (DIC) detailreiche Bilder von der allgemeinen Morphologie der Proben, und darüber hinaus noch mit deutlich geringerer Photonendosis. Während den Hellfeld-Techniken die erforderliche molekulare Spezifität fehlt, ist es jedoch die gute Praxis, immer wieder mal Hellfeldbilder aufzunehmen, um im Verlauf des Experiments morphologisch den Zustand der lebenden Zellen und Gewebe besser kontrollieren und beurteilen zu können.
Glossar
- Grenzbereich für das Imaging in tieferen Schichten
- Das ist die ungefähre Distanz entlang der Z-Achse, über die das jeweilige System mit dem Objektiv in tiefere Schichten der Probe „hineinfokussieren“ kann, um noch optische Schnitt-Bilder mit angemessener Qualität und ausreichendem Signal-Rausch-Verhältnis zu liefern. Dieser Wert kann sehr variabel sein und hängt stark von den optischen Eigenschaften der Probe, der Art der Probenkammer sowie der (Fluoreszenz-) Färbung ab.
- Kompatible Mikroskopstative
- Hier wird angezeigt, welche Nikon Mikroskopstative mit dem jeweiligen System kompatibel sind.
- Sehfeld
- Das Sehfeld eines Mikroskop-Systems, auch mit Sehfeldzahl bezeichnet ist der Durchmesser (in mm) des reellen Zwischenbildes („imaging area“) bei einer nominalen Vergrößerung von 1x.
- Unterstützt Imaging mit Video-Rate
- Als „Video-Rate“ wird traditionell eine Aufnahmegeschwindigkeit von etwa 30 Bildern pro Sekunde (FPS) definiert. Die optimale Bildrate hängt von der genauen Anwendung ab und kann schneller oder langsamer als 30 FPS sein. EM-CCD-Kameras können typischerweise Bilder mit bis zu 60 FPS (Vollbild) und sCMOS-Kameras mit bis zu 40-100 FPS (Vollbild) aufnehmen.
- Unterstützte Mikroskopiertechniken
- Dies bezieht sich auf die verschiedenen Mikroskopiertechniken, die von den jeweiligen Systemen bereitgestellt werden. Beachten Sie, dass fast alle Mikroskopiertechniken, die vom inversen Mikroskop ECLIPSE Ti2-E unterstützt werden, auch dann genutzt werden können, wenn es als Basis für jedes der anderen in dieser Tabelle aufgeführten Systeme verwendet wird.
- Home
- Anwendungen
- Biowissenschaften
- Live-Cell-Imaging