- de Change Region
- Global Site
- Home
- Anwendungen
- Biowissenschaften
- Deep Learning in der Mikroskopie
Deep Learning in der Mikroskopie
Methoden der künstlichen Intelligenz (KI) werden für die Analyse von Mikroskopie-Daten immer beliebter. Deep Learning (DL) ist eine Methode des maschinellen Lernens (ML) und verwendet zum Lernen künstliche neuronale Netzwerke (KNN). KNNs werden so genannt, weil sie Ähnlichkeiten mit unserem eigenen Gehirn haben – jedes “Neuron” im Netzwerk erhält Eingaben von mehreren anderen verbundenen “Neuronen”. Der Trainingsprozess ändert die Gewichtung dieser Verbindungen, um die Ergebnisse, die von einer gegebenen Eingabe (Input) erwartet werden, am besten vorherzusagen (Output).
Produkte für Deep Learning in der Mikroskopie
Lösungswege mit DL können vorteilhaft auf eine Vielzahl von Bildanalyseaufgaben angewendet werden. Die NIS.ai-Softwaremodule für die Nikon NIS-Elements-Software nutzen DL für verschiedene Bildanalyse-Fragestellungen. NIS.ai-Module können mit anderen Software-Tools als Teil automatisierter Imaging- und Analyse-Pipelines kombiniert werden, indem unser JOBS-Experiment-Wizard bzw. das Modul General Analysis 3 (GA3) verwendet wird.
●: enthalten, ⚬: option
| Autosignal.ai | Clarify.ai | Convert.ai | Denoise.ai | Enhance.ai | Segment.ai | |
|---|---|---|---|---|---|---|
| Funktion | Ermittelt automatisch optimale Laserleistung und Gain-Einstellung für das Imaging mit konfokalem Punktscanner. | Entfernt automatisch unscharfe out-of-focus-Fluoreszenzsignale aus Weitfeld-Fluoreszenzbildern. | Sagt für einen definierten Imaging- Kanal Bilder von Strukturen voraus, die anhand von Mustern in einem Durchlichtkanal identifiziert wurden. | Entfernt automatisch Rauschen aus Bildern. | Prognostiziert ein Bild mit verbessertem Signal-Rausch-Verhältnis aus einem Bild mit schwachem Signal. | Segmentiert komplexe Bildmerkmale basierend auf Mustern, die durch benutzergeführtes Training identifiziert wurden. |
| Vorheriges Training notwendig | nein | nein | ja | nein | ja | ja |
| Kompatible Nikon-Systeme | Konfokale Mikroskope AX / AX R | alle | alle | Konfokale Mikroskope AX R und Systeme mit CMOS-Kamera(s) | alle | alle |
| Softwarepakete | Autosignal.ai | Clarify.ai | Convert.ai | Denoise.ai | Enhance.ai | Segment.ai |
| NIS-Elements C | Optional | Optional | Optional | Optional | Optional | Optional |
| NIS-Elements AR | no | Optional | Optional | Optional | Optional | Optional |
| NIS-Elements AR ML | no | Optional | Optional | Optional | Optional | Optional |
| NIS-Elements AR Passiv | no | Optional | Optional | Optional | Optional | Optional |
| NIS.ai-Modul | yes | Optional | Optional | ja | Optional | Optional |
| Gebündelt mit Modulen für Dekonvolution | nein | ja | no | nein | nein | nein |
Zugehörige Literatur
Diskussion über Deep Learning in der Mikroskopie
Die Vorteile und Grenzen von Deep Learning für die Mikroskopie
DIC-, Ground Truth- und Convert.ai-Rekonstruktionsbilder eines Sphäroids in Kultur. Convert.ai war in der Lage, die Position und Form der Strukturen, die durch den Fluoreszenzfarbstoff markiert worden wären angemessen aus dem DIC-Bild zu rekonstruieren, wenn auch nicht jedes Detail.
Deep Learning-basierte Algorithmen und Software sind eine viel versprechende Unterstützung für die Bildanalyse in der optischen Mikroskopie, jedoch ist es wichtig zu verstehen, dass solche Ansätze kein “Allheilmittel” sind, sondern vielmehr eine neuartige Herangehensweise an komplexe Fragestellungen bei der Bilddatenauswertung. Deep Learning (DL)-Algorithmen, die Korrelationen zwischen Datenmerkmalen ableiten können (die in den Bilddaten nicht selten tief verborgen sind oder unwillkürlich auftreten), sind für nicht wenige Mess- und Darstellungsaufgaben besser geeignet als klassische Ansätze. Als der überwiegende Vorteil der künstlichen Intelligenz (KI) für die Mikroskopie erscheint die Beschleunigung der Bildanalyse.
DL-Algorithmen für das überwachte Lernen, wie sie von Nikons NIS.ai-KI-Modulen verwendet werden, erfordern umfangreiche Daten zum Trainieren des neuronalen Netzwerks. Während Nikon einige KI-Module vorab anlernt, darunter Denoise.ai, Clarify.ai und Autosignal.ai, müssen andere unter Verwendung der spezifischen Aufnahmebedingungen für bestimmte Experiment-/Präparat-Situationen trainiert werden. Dieser Prozess muss äußerst gewissenhaft durchgeführt werden und kann langwierig sein. Aber sobald er abgeschlossen ist, wird eine schnelle Analyse neuer Daten möglich. Es ist jedoch wichtig zu verstehen, dass die Bildergebnisse nicht (exakt) die “Ground-truth” darstellen. Letztlich schränkt dies die entsprechenden Anwendungen zu einem gewissen Grad ein.
Hier ein geeignetes Anwendungs-Beispiel: Das Modul Convert.ai- kann darauf trainiert werden, das DAPI-Kernfärbungsmuster aus Durchlichtbildern (z.B. DIC) vorherzusagen. Diese vorhergesagten “DAPI-Bilder” sind sehr geeignet für Zellzählungen, die anhand der korrekten Lokalisierung des “DAPI-Signals” relativ einfach und schnell durchführbar sind. Die Verwendung vorhergesagter “DAPI-Bilder” für eine Quantifizierung - z.B. für Intensitätsmessungen der “Fluoreszenz” in den Zellkernen - wäre jedoch unangemessen. Während DL für die genaue Vorhersage der DAPI-Kernfärbung und ähnlicher, vergleichsweise grober Färbungsmuster für das reine Zählen sehr zuverlässig ist, sind diese erzeugten “Fluoreszenzbilder” keine gute Wahl für quantitative Analysen feiner Details und auch seltener Ereignisse (“rare events”), von denen letztere in den Trainingsbilddaten von Anfang an nicht gut repräsentiert wären oder wahrscheinlich sogar übersehen würden.
Imaging und Analysen mit Deep Learning sinnvoll nutzen
Schnittpräparat, Darm (Maus), Fluoreszenzmarkierung, aufgenommen mit dem konfokalen Mikroskop Nikon AX-R-Resonanz-Scanner. Objektiv: 20X WI, NA 0,95
Ein Vorteil von DL-Methoden ist, dass sie schnell verschiedene und komplexe Aufgaben erledigen können. Dies ermöglicht das Aneinanderfügen von DL-Methoden, um komplizierte mehrteilige Analysen relativ schnell hintereinander durchzuführen, sogar während Bilder aufgenommen werden, wenn die Zeit begrenzt ist.
Ein einfaches Beispiel: das Modul Autosignal.ai stellt bei unserem konfokalen Mikroskop AX / AX R innerhalb weniger Sekunden automatisch die jeweils optimale Laserleistung und elektronische Signal-Verstärkung (“gain”) ein. Es ist unwahrscheinlich, dass ein klassischer Ansatz für diesen fundamentalen Schritt bei der Einstellung für ein optimales Bild in so kurzer Zeit eine zuverlässige Lösung liefern würde. Aber ein paar Sekunden können einen signifikanten Unterschied machen, wenn es um so wichtige Faktoren wie z.B. Photobleaching geht.
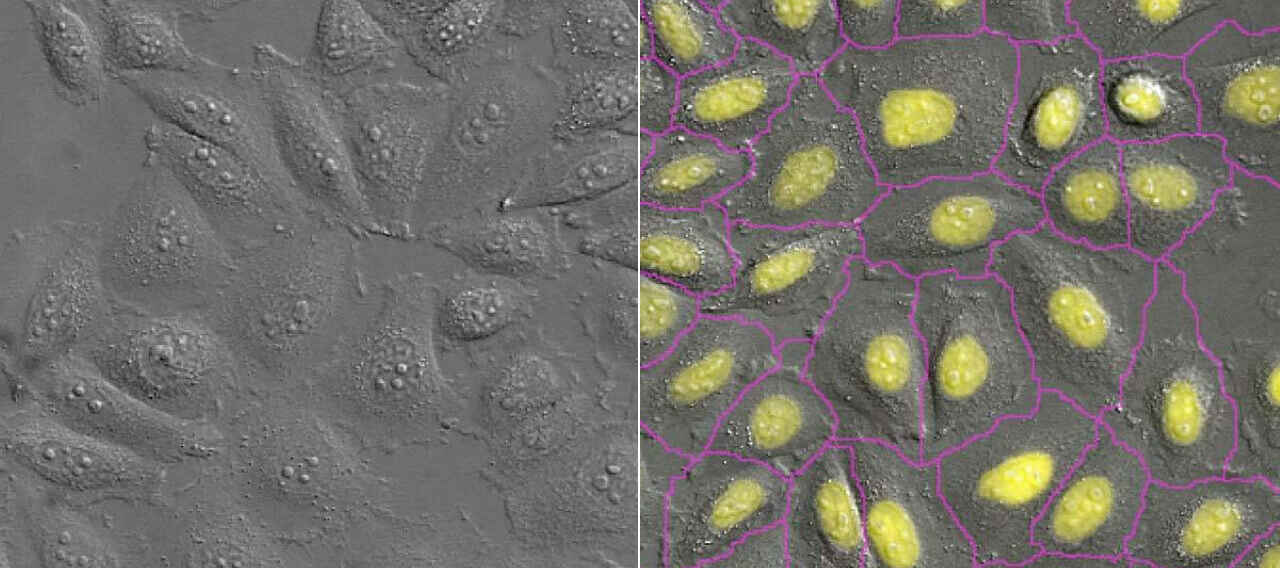
Das Denoise.ai-Modul ist ähnlich schnell und entfernt Rauschen aus Bildern nahezu in Echtzeit. Dadurch ergibt sich eine synergetische Kombination mit Autosignal.ai – die Bedingungen für die Darstellung optimaler Bilder können innerhalb von Sekunden per Knopfdruck eingestellt werden. Andere KI-Module können in ähnlicher Weise angefügt werden, beispielsweise Segment.ai für die schnelle Segmentierung von Bildmerkmalen, wie im Video gezeigt.
NIS.ai-Module können in bestehende Imaging- und Analyse-Pipelines mit unserer NIS-Elements-Software integriert werden. Dies ist vorteilhaft für die Optimierung der Effizienz von High-Content-Experimenten. Das Modul General Analysis 3 (GA3) wird verwendet, um benutzerdefinierte Messroutinen zu erstellen, und ermöglicht es, NIS.ai DL-basierte und klassische Analysen zusammen in derselben Pipeline anzuwenden. NIS.ai-Module können auch mit dem JOBS-Experiment-Wizard in mikroskopische Imaging- Routinen eingebettet werden – und das Besondere: Ergebnisse, die mit einem NIS.ai-Modul in JOBS erzielt werden, können dazu verwendet werden, den Verlauf des Experiments zu steuern. Beispielsweise kann man basierend auf Analyseergebnissen automatisch gezielte Bildaufzeichnungen auslösen – eine geeignete und sehr aussichtsreiche Chance, um seltene Ereignisse im Präparat gezielt zu finden.
Deep Learning für Live Cell Imaging
Vergleich Rohbilder und Denoise.ai-Bilder ohne Rauschen. Zeitraffer-Aufnahme mit Nikon Konfokal-Mikroskop A1 R mit Resonanzscanner
Drosophila sp. Larve. Zur Verfügung gestellt von Amicia Elliot, PhD, NIH/NIMH, Bethesda MD. Aufgenommen im Quantitative Fluorescence Microscopy Course 2019.
Aufnahme mit: Resonanzscanner, Bildgröße 1024 x 512, Scangeschwindigkeit 30 fps, Scan-Zoom x 0,72, kein Averaging, Objektiv Plan Apo 4X
Einer der größten Vorteile vieler NIS.ai-KI-Module ist die Reduzierung der Photonendosis für die Proben. Denoise.ai und Clarify.ai liefern unmittelbar klarere Bilder, ohne dass zusätzliche Bilder für notwendige Signale gescannt oder erfasst werden müssen. Convert.ai ermöglicht die Vorhersage von “Fluoreszenz-Bildern” aus Durchlichtbildern (z. B. DIC), wodurch in entsprechend geeigneten Fällen die Notwendigkeit einer Fluoreszenz-Mikroskopie sogar vollständig entfällt. Enhance.ai kann Bilder mit einem verwertbaren, höheren Signal aus Bild-Daten mit niedrigem Signal vorhersagen, wodurch die für die Anregung erforderliche Photonendosis minimiert wird.
Die Fluoreszenzmikroskopie erfordert im Allgemeinen eine viel höhere Beleuchtungsintensität als Durchlichtverfahren, hat ein begrenztes Photonenbudget und ist aber dennoch häufig die Methode der Wahl für das Live-Cell-Imaging, insbesondere in der Forschung. Die Reduzierung der Photonendosis ist für die meisten Mikroskopie-Anwendungen ist immer gewünscht, sie ist jedoch für die Fluoreszenzmikroskopie an lebenden Zellen unerlässlich, und stets ernsthaft in Betracht zu ziehen ist.
Die Aussichten von KI und Deep Learning in der Mikroskopie
Das Training von NIS.ai-Modulen ist unkompliziert. Die Software bietet eine Echtzeitansicht des Trainingsfortschritts (gezeigt ist Enhance.ai).
Vergleich der Low-Signal-DAPI-Bilddaten (links) mit dem vorhergesagten Bild nach der prognostizierten Signal-Restauration durch Enhance.ai (rechts)
Die Anwendung von KI und Deep Learning in der Mikroskopie befindet sich noch in einem relativ frühen Stadium, und es werden ständig neue Forschungsergebnisse und Anwendungen veröffentlicht, die diese Analysemöglichkeiten vorantreiben. Angesichts des Potenzials von DL-Methoden und der Tatsache, wie schnell solche Methoden zum Mainstream werden, ist es wichtig, dass die KI- und Mikroskopie-Community klare Richtlinien und Qualitätsmerkmale für geeignete Anwendungen entwickelt, die es “Laien” ohne detaillierten Background in KI und Programmierung zu ermöglichen, diese Tools für Peer-Reviewed-Forschung sicher anzuwenden.
Die vortrainierten KI-Module von Nikon, zu denen derzeit Clarify.ai und Denoise.ai gehören, lassen keine manuelle Anpassung mehrdeutiger Einstellungen zu, wie z.B. der “Intensität” der Analyse, wodurch die Möglichkeit vermieden wird, dass subjektive Betrachtungsweisen vom Benutzer die Ergebnisse beeinflussen. Jeder, der Clarify.ai oder Denoise.ai verwendet, verwendet dasselbe vortrainierte Netzwerk wie jeder andere Benutzer.
NIS.ai-Module, die trainiert werden müssen, einschließlich Convert.ai, Enhance.ai und Segment.ai, enthalten eine intuitive grafische Benutzeroberfläche für das Trainieren des neuronalen Netzwerks. Der Benutzer kann den Fortschritt des Trainings überwachen, einschließlich einer Echtzeitansicht der Anzahl der Iterationen und möglicher Fehler in den Lerndaten.
Glossar
- Funktion
- Zusammenfassung der Funktion des jeweiligen NIS.ai-Software-Moduls
- Kompatible Nikon-Systeme
- Gibt an, welche Nikon-Mikroskope/Systeme kompatibel sind. Dies setzt voraus, dass das Mikroskop/System für die Aufnahme digitaler Bilder ausgerüstet ist.
- Softwarepakete
- Liste der käuflich erhältlichen NIS-Elements-Softwarepakete und -Module. Einzelne NIS.ai-Module können in den gelisteten Software-Paketen und Modulen wie angegeben enthalten oder für sie optional verfügbar sein.
- Vorheriges Training notwendig
- Das Training eines Deep Learning neuronalen Netzwerks für die Bildanalyse wird beim sog. “überwachtem Lernen” häufig mit gepaarten Bilddaten durchgeführt: einem Eingangsbild (das nach dem Training als sog. Input zu analysieren ist) und sog. Ground-truth-Bilddaten desselben Bereichs, aus denen die gewünschte Zielsetzung für das angestrebte Analyseergebnis hervorgeht. Die Gewichtungen der Knotenpunkte innerhalb des neuronalen Netzwerks werden dann sukzessiv angepasst, um Fehler zwischen dem vorhergesagten Output-Bild (-Ergebnis) und dem Ground-Truth-Bild zu minimieren.
- Home
- Anwendungen
- Biowissenschaften
- Deep Learning in der Mikroskopie