- fr Change Region
- Global Site
- Accueil
- Ressources
- Focus On
- Virology
Focus sur la Virologie
Autorisation du film par Rudolph Reimer, Institut Heinrich Pette, Institut Leibniz de virologie expérimentale
Technologie de capture d'images et de quantification de l'activité virale en temps réel
La taille extrêmement petite des virus (5 à 300 nm) rend leur structure et leur fonction difficiles à étudier. Au cours des derniers siècles, de nombreux virus ont été découverts et des vaccins contre eux mis au point, mais c'est l'invention du microscope électronique en 1931 qui a pour la première fois éclairé leur structure complexe. Depuis lors, les techniques de microscopie optique telles que l’imagerie en fluorescence se sont développées pour jouer un rôle important dans la recherche virale, permettant d'étudier l'activité virale dans les systèmes vivants. Nikon est l'un des principaux fabricants de systèmes avancés de microscopie optique pour la capture d'images de l'activité virale en temps réel avec une haute résolution et un débit élevé. Compte tenu de l'importance croissante d'une analyse rapide et fiable de l'activité virale, Nikon s'engage plus que jamais dans le développement et la prise en charge de systèmes avancés pour de tels travaux.
Imagerie de cellules vivantes
Un large éventail de capacités de Nikon contribue à une imagerie réussie à long terme et en accéléré des processus viraux avec un photoblanchiment minimal, incluant la microscopie confocale et à large champ de haute vitesse, la technologie logicielle basée sur l'intelligence artificielle (IA) et un système de verrouillage et mise au point en temps réel (Perfect Focus System, PFS).
Le microscope inversé à large champ ECLIPSE Ti2 est une plateforme puissante pour l’imagerie de cellules vivantes en virologie. Son champ de vision (FOV) inégalé de 25 mm capture les changements et les réactions des cellules et des virus sur une large zone en un seul coup. Le système de mise au point parfaite (PFS) empêche la dérive focale, gardant les cellules en mouvement et les virus constamment mis au point et prenant ainsi en charge l’imagerie de haute résolution lors d'acquisitions à grande vitesse et à long terme.
Les microscopes confocaux AX/AX R, CSU-W1 SoRa et Crest X-Light V3 ont un FOV (jusqu'à 25 mm) qui est presque le double de celui des microscopes confocaux conventionnels. Cela permet de collecter plus de données dans une seule image, ce qui se traduit par une plus grande efficacité. L'observation en temps réel des activités virales dans les cellules vivantes est également possible grâce à la microscopie TIRF (Total Internal Reflection Fluorescence), dont les modules peuvent être installés sur le microscope inversé Ti2. TIRF permet l'étude des processus à proximité de la membrane cellulaire avec un rapport signal/bruit exceptionnellement élevé, comme le mécanisme d'entrée virale dans les cellules et son impact sur les interactions cellule-cellule. Une résolution améliorée est obtenue sur le disque tournant CSU-W1 SoRa confocal en utilisant le concept de réassignation optique des photons. Le disque rotatif Crest X-Light V3 confocal fournit un éclairage homogène sur un large champ de vision de 25 mm.
La phototoxicité et le photoblanchiment des sondes fluorescentes utilisées pour colorer les virus peuvent être minimisés à l'aide du scanner résonant de haute vitesse de l'AX R. La puissance du microscope confocal AX R réside dans la combinaison du scanner résonnant de haute définition et du large champ de vision de 25 mm, qui offre un débit élevé. Toutes les solutions d’imagerie en direct de Nikon intègrent NIS-Elements, une plateforme logicielle unifiée d'acquisition et d'analyse qui prend désormais en charge les modules d'apprentissage en profondeur NIS.ai de Nikon, notamment Clarify.ai, Denoise.ai et Enhance.ai.
Clarify.ai supprime automatiquement le flou des images en fluorescence à champ large à l'aide d'un réseau neuronal préformé. Denoise.ai supprime le bruit de tir de Poisson des données confocales résonnantes, en temps réel ou post-acquisition, et également en utilisant un réseau de neurones pré-entraîné. Lorsqu'il est combiné avec le microscope confocal AX R, Denoise.ai, ilpermet aux utilisateurs d'acquérir rapidement des images de caractéristiques de faible rapport signal/bruit qui nécessiteraient normalement des temps de séjour plus longs pour être suffisamment résolus. Enhance.ai est utile pour l’imagerie en fluorescence à faible luminosité des virus, ce qui est nécessaire pour les marqueurs de détection photosensibles présents en faible nombre. Le réseau neuronal est formé à l'aide de données d'images de rapport signal/bruit apparié haut et faible, ce qui lui permet de prédire rapidement les versions signal / bruit élevées des images d'entrée suivantes.
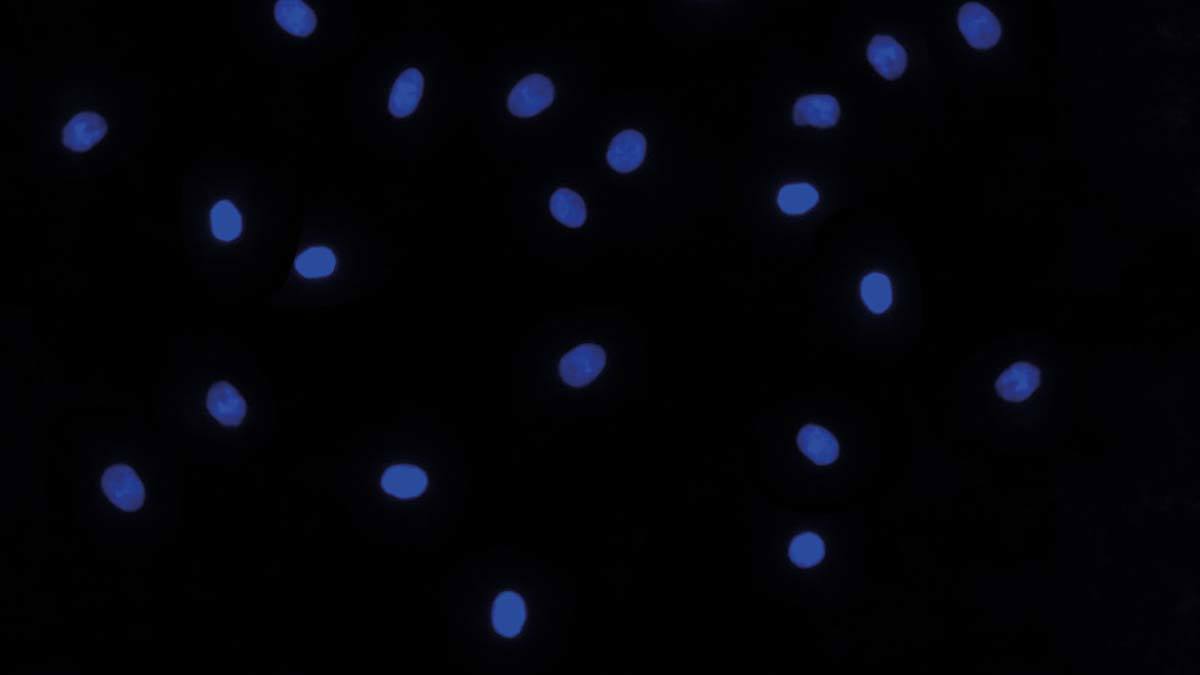

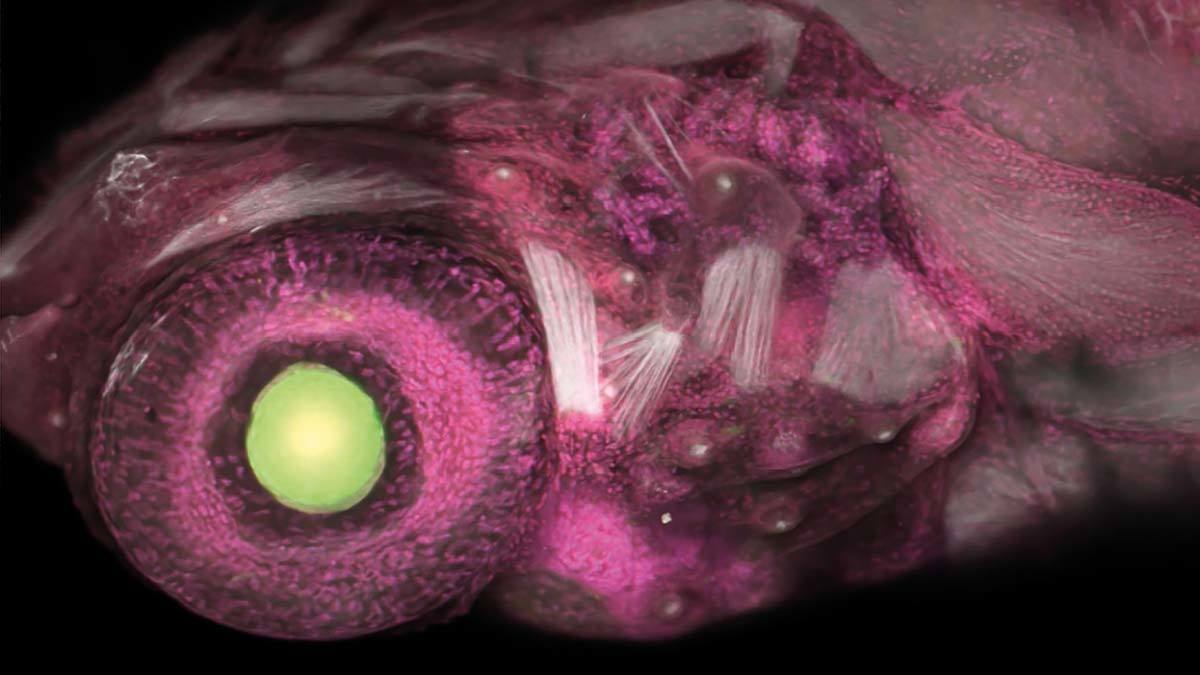
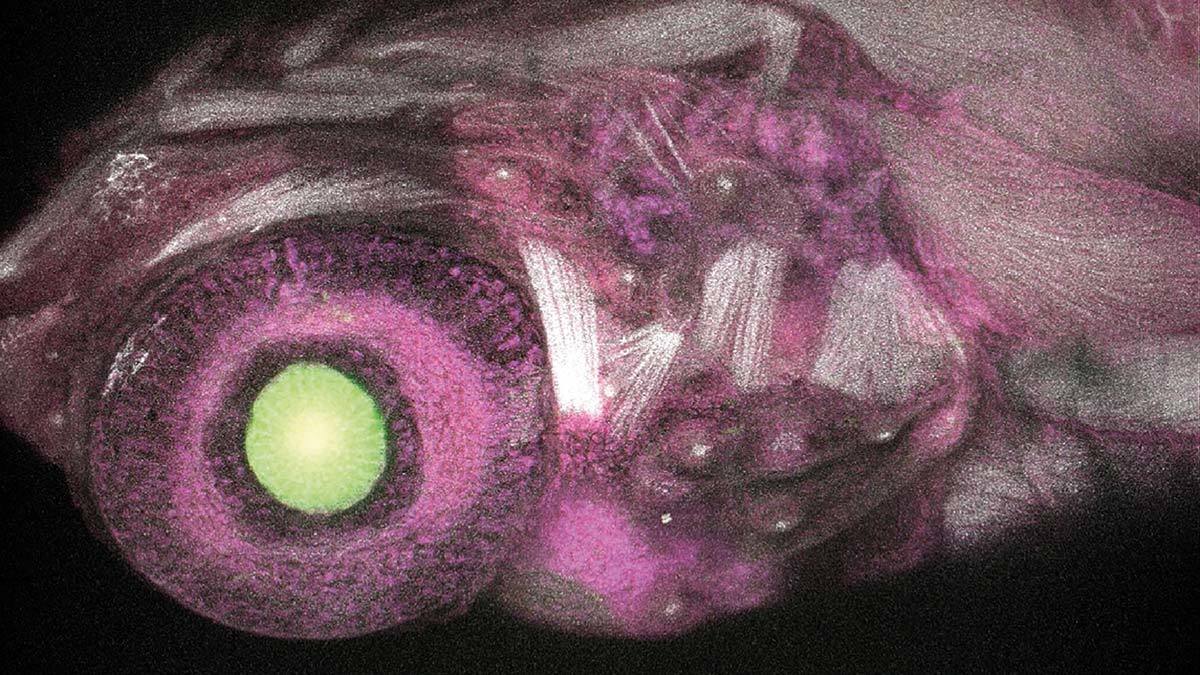
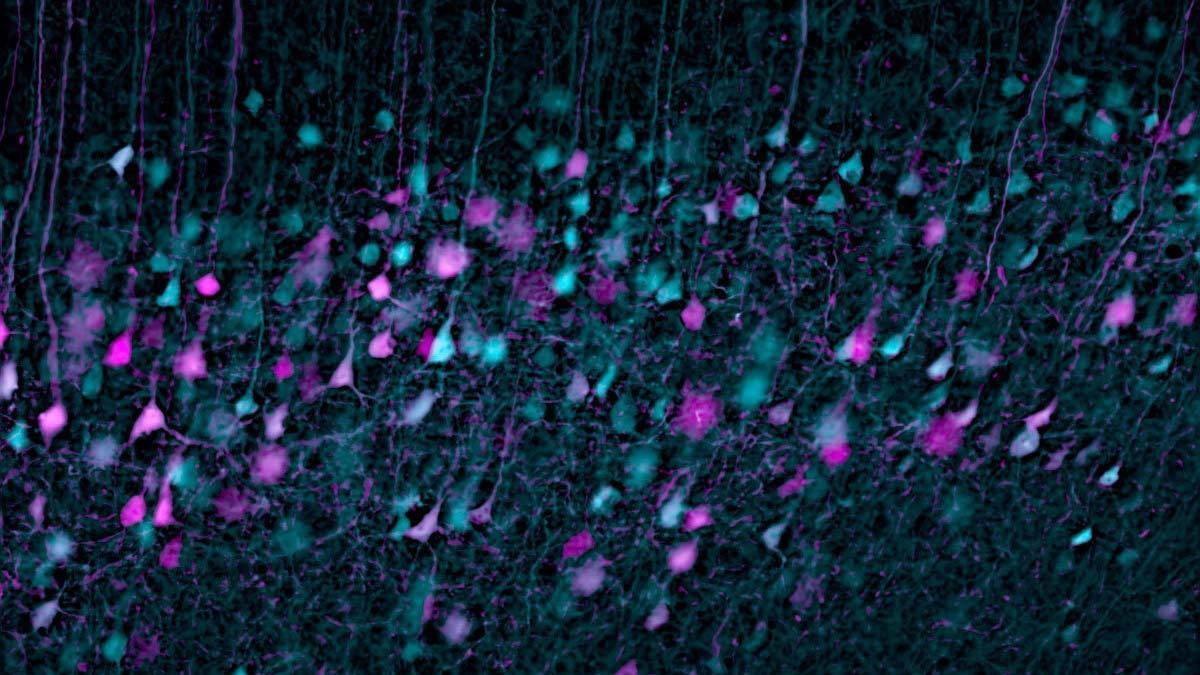
Étude de cas:
Le transport et la propagation du virus de l’herpès alpha dans les cultures neuronales ont été étudiés par l'équipe du Dr Lynn Enquist de l'Université de Princeton, aux États-Unis, en utilisant une plateforme Nikon de capture d'images à champ large de cellules vivantes (le microscope inversé ECLIPSE Ti contrôlé par le logiciel NIS-Elements). Leur travail applique la capture d'images de cellules vivantes en temps réel et pendant la nuit de virus exprimant des fusions de protéines fluorescentes pour illuminer les assemblages viraux pendant l'infection des neurones primaires. Leur approche minimise également l'impact de la capture d'images en fluorescence sur le virus.
(Taylor, M.P. et al. (2013) J. Vis. Exp. (78), e50723, doi:10.3791/50723).
Dépistage de haut contenu
Lorsqu'un dépistage clinique à grande échelle, comme les tests de médicaments et de virus, est nécessaire, une quantité considérable de données d'images multidimensionnelles est produite en peu de temps. Nikon fournit une plateforme de criblage cellulaire de haut contenu (BioPipeline Live), un outil de programmation visuelle (JOBS) et un outil d'analyse de haute performance (GA3) pour rationaliser et automatiser votre flux de travail de capture d'images virales.
La plateforme de capture d’images de haut contenu en cellules vivantes de Nikon, BioPipeline Live, constitue une excellente solution pour la capture d’images en direct entièrement automatisée dans plusieurs plaques. Utilisant toutes les fonctionnalités du microscope inversé ECLIPSE Ti2, le BioPipeline Live comprend une automatisation robotique pour une capture d'images rapide et un échange de jusqu'à 44 plaques!
L'outil General Analysis 3 (GA3) permet la mise en œuvre simple d'analyses de post-traitement complexes. Les images incluent des métadonnées complètes, même pour des paramètres tels que les conditions environnementales, qui peuvent être cruciales pour les études virales. En plus de toutes les fonctions d'analyse et de traitement fournies par NIS-Elements, le module JOBS offre la possibilité de créer des protocoles d'expérimentation personnalisés, y compris des flux de travail conditionnels. Cela permet une acquisition et une analyse entièrement automatisées de plusieurs plaques. Cela comprend la visualisation immédiate des données de mesure, puits par puits, et via une carte thermique pour l'observation des tendances et une analyse plus approfondie. Le débit avec diverses tâches peut être augmenté ainsi que la viabilité des échantillons à l'aide des modules NIS.ai basés sur l'apprentissage en profondeur disponibles pour NIS-Elements. Par exemple, Segment.ai applique un réseau neuronal pour segmenter automatiquement vos images (une fois entraînées). JOBS peut rationaliser et automatiser l'ensemble de la plateforme, en intégrant des fonctions telles que le contrôle des appareils, l'acquisition d'images, le traitement et l'analyse sans avoir besoin de connaissances avancées en programmation.
Étude de cas:
Le groupe de recherche du Dr Vibor Laketa à l’université de Heidelberg, en Allemagne, a récemment mis au point un test semi-quantitatif d’anticorps anti-SARS-CoV-2 à utiliser sur des sérums humains. Ce test basé sur un microscope est démontré à l'aide d'un microscope à champ large Nikon ECLIPSE Ti2 contrôlé par le module JOBS du logiciel NIS-Elements pour acquérir automatiquement des images dans des plaques de 96 puits. Ce nouveau test démontre une sensibilité et une spécificité améliorées par rapport à un test diagnostique standard basé sur ELISA, et avec un débit élevé, ce qui est nécessaire pour les grands programmes de dépistage.
(Pape, C. et al. (2020) bioRxiv (préimpression doi: https://doi.org/10.1101/2020.06.15.152587)).
Super-résolution
Capture d'images en super-résolution de la structure et de l'activité virales
Les particules virales varient considérablement en taille, s'étendant sur environ 5 à 300 nm de diamètre, ce qui va de l’au-delà à proche de la limite de résolution de la microscopie à fluorescence conventionnelle. Pendant des centaines d'années, la microscopie optique ne pouvait résoudre les détails cellulaires qu'à environ 200 nm en XY et 500 nm en Z, mais de nouvelles techniques de super-résolution ont réussi à résoudre les détails biologiques à environ 20 nm en XY et 50 nm en Z. Cela permet d'étudier des micro-organismes petits et, parfois, rares (y compris des virus) avec des détails de niveau presque moléculaire. Ces techniques de super-résolution aident à combler le fossé entre la microscopie à fluorescence conventionnelle et la microscopie électronique.
Nikon propose une gamme de systèmes de super-résolution pour différentes applications de capture d'images. La microscopie à illumination structurée (SIM) fournit deux fois la résolution d'un microscope à champ large typique en X, Y et Z tout en maintenant une vitesse d'acquisition élevée et des exigences de préparation d'échantillon flexibles. La microscopie de reconstruction optique stochastique (STORM) offre le plus grand niveau de détail - jusqu'à 10 fois plus de résolution que les techniques conventionnelles.
SIM utilise le concept de mélange de fréquences pour identifier les informations de fréquence de super-résolution dans l'image. Cela fonctionne en éclairant l'échantillon avec divers modèles de fréquence spatiale connue et en comparant la façon dont l'image est modulée par chaque modèle, avec une analyse ultérieure révélant des informations de fréquence de super-résolution qui ne sont pas directement observables et en les utilisant pour calculer l'image finale. La vitesse d'acquisition pour le N-SIM S est jusqu'à 15 images par seconde, ce qui est activé en utilisant un modulateur de lumière spatiale (SLM) pour la modulation de modèle, et particulièrement utile pour visualiser la progression rapide de l'activité virale dans les cellules vivantes. Le N-SIM S peut être implémenté sur le même support de microscope qu'un système confocal. Une telle approche corrélative permet une capture d'images confocales rapide de large champ de vision, réservant l'acquisition de super-résolution pour les régions d'intérêt.
Le microscope à super-résolution N-STORM repousse les limites de la résolution optique en « activant » seulement un petit nombre de fluorophores non chevauchants dans un échantillon. Cela permet d'identifier la position centroïde d'événements à émission unique avec une grande précision. La répétition de ce processus sur de nombreuses trames de captures d'images permet de reconstruire l'image complète à une résolution sans précédent, à environ 20 nm dans les directions XY et 50 nm dans les directions Z. La lentille à immersion 100x en silicone de Nikon (NA = 1,35) permet des performances 3D supérieures dans les cultures de cellules vivantes et les échantillons plus épais, car elle corrige mieux les aberrations sphériques dans les milieux aqueux.
Comme pour tous les microscopes Nikon, le logiciel NIS-Elements est fondamental pour une capture d’images réussie, simplifiant considérablement l’acquisition et l’analyse des images en super-résolution.
Étude de cas:
Le Centre d'excellence Nikon (CoE) de l'Institut Heinrich Pette (HPI), Hambourg, Allemagne, est un centre de recherche et d'éducation sur les principes fondamentaux et les progrès de la microscopie pour l'étude des virus pathogènes humains. Une étude de HPI a démontré les capacités de la technologie multicolore 3D STORM pour l'imagerie super-résolution de l'infection par le virus de l'hépatite C (VHC). STORM a permis de visualiser la distribution spatiale du noyau structurel et de la protéine d'enveloppe E2 du VHC dans de petites gouttelettes lipidiques sur la membrane du réticulum endoplasmique dans les cellules infectées. Ces zones de colocalisation ont un diamètre d'environ 100 nm et représenteraient des sites d'assemblage viraux.
(Eggert, D. et al. (2014) PloS ONE 9(7): e102511. Doi:10.1371/journal.pone.0102511).
- Accueil
- Ressources
- Focus On
- Virology